Topik ini menjelaskan solusi yang menggunakan mesin vektor TairVector untuk melakukan pencarian aproksimasi struktur molekul senyawa atau obat.
Latar belakang
Pengambilan vektor memainkan peran penting dalam penemuan obat berbasis AI. Di bidang ini, vektor merepresentasikan senyawa dan obat. Perhitungan kemiripan dalam ruang vektor membantu memprediksi dan mengoptimalkan interaksinya, sehingga memungkinkan penyaringan cepat senyawa dan obat dengan interaksi kuat serta mempercepat pengembangan obat baru. Selain itu, pengambilan vektor meningkatkan akurasi dan efisiensi penyaringan obat, memberikan metode penelitian yang lebih efisien dan tepat bagi peneliti medis.
Dibandingkan layanan pengambilan vektor tradisional, TairVector menyimpan seluruh data di memori dan mendukung pembaruan indeks secara real-time untuk latensi baca-tulis yang lebih rendah. Perintah kueri tetangga terdekat, seperti TVS.KNNSEARCH, memungkinkan Anda mengambil secara efisien k struktur molekul paling mirip dari database dengan menyesuaikan nilai k. Hal ini mengurangi risiko kegagalan proyek akibat kesalahan atau kelalaian manusia.
Ikhtisar solusi
Bagan alir berikut menunjukkan prosesnya.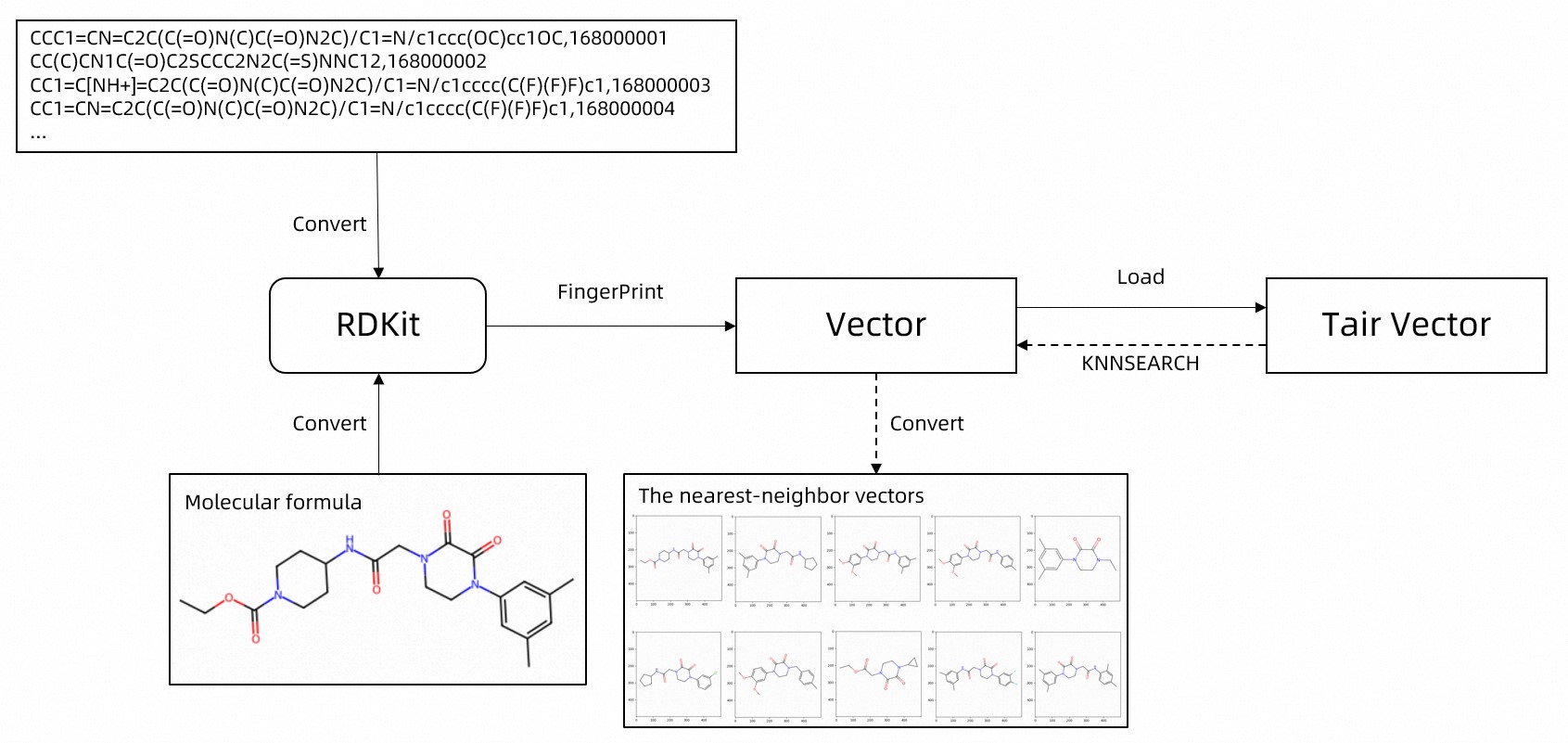
Unduh set data struktur molekul.
Contoh ini menggunakan data uji dari set data sumber terbuka PubChem yang berisi 11.012 baris data. Data dari tautan unduhan berada dalam format Simplified Molecular Input Line Entry System (SMILES). Contoh berikut menampilkan dua kolom: formula kimia dan ID unik.
CatatanDalam proyek dunia nyata, Anda dapat menulis lebih banyak data untuk menguji performa pengambilan Tair pada tingkat milidetik.
CCC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1ccc(OC)cc1OC,168000001 CC(C)CN1C(=O)C2SCCC2N2C(=S)NNC12,168000002 CC1=C[NH+]=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1cccc(C(F)(F)F)c1,168000003 CC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1cccc(C(F)(F)F)c1,168000004Jika Anda mengunduh data dari website resmi, datanya berada dalam format Structure-Data File (SDF). Anda harus mengonversinya ke format SMILES menggunakan kode berikut:
Hubungkan ke instans Tair. Untuk informasi selengkapnya, lihat fungsi
get_tairdalam kode contoh.Buat indeks vektor di Tair untuk menyimpan struktur molekul. Untuk informasi selengkapnya, lihat fungsi
create_indexdalam kode contoh.Tulis data struktur molekul sampel. Untuk informasi selengkapnya, lihat fungsi
do_loaddalam kode contoh.Ekstrak fitur vektor dari data struktur molekul menggunakan pustaka RDKit. Kemudian, gunakan perintah mesin vektor TVS.HSET untuk menyimpan ID unik, informasi fitur, dan formula kimia di Tair.
Lakukan pencarian kemiripan struktur molekul. Untuk informasi selengkapnya, lihat fungsi
do_searchdalam kode contoh.Ekstrak fitur vektor molekul kueri menggunakan pustaka RDKit. Kemudian, gunakan perintah mesin vektor TVS.KNNSEARCH untuk mengkueri indeks tertentu di Tair guna menemukan struktur molekul paling mirip.
Contoh kode
Contoh ini menggunakan Python 3.8. Instal pustaka dependensi berikut terlebih dahulu: pip install numpy rdkit tair matplotlib.
import os
import sys
from tair import Tair
from tair.tairvector import DistanceMetric
from rdkit.Chem import Draw, AllChem
from rdkit import DataStructs, Chem
from rdkit import RDLogger
from concurrent.futures import ThreadPoolExecutor
RDLogger.DisableLog('rdApp.*')
def get_tair() -> Tair:
"""
Hubungkan ke instans Tair.
* host: Titik akhir instans Tair.
* port: Nomor port instans Tair. Nilai default adalah 6379.
* password: Kata sandi akun default untuk instans Tair. Untuk menghubungkan dengan akun kustom, gunakan format 'username:password'.
"""
tair: Tair = Tair(
host="r-bp1mlxv3xzv6kf****pd.redis.rds.aliyuncs.com",
port=6379,
db=0,
password="Da******3",
)
return tair
def create_index():
"""
Buat indeks vektor untuk menyimpan struktur molekul:
* Nama indeks dalam contoh ini adalah "MOLSEARCH_TEST".
* Dimensi vektor adalah 512.
* Metrik jarak adalah L2.
* Algoritma indeks adalah HNSW.
"""
ret = tair.tvs_get_index(INDEX_NAME)
if ret is None:
tair.tvs_create_index(INDEX_NAME, 512, distance_type=DistanceMetric.L2, index_type="HNSW")
print("create index done")
def do_load(file_path):
"""
Masukkan path ke set data struktur molekul. Metode ini secara otomatis mengekstrak fitur vektor struktur molekul (smiles_to_vector) dan menulis data ke Tair Vector.
Metode ini juga memanggil fungsi parallel_submit_lines, handle_line, smiles_to_vector, dan insert_data.
Data disimpan di Tair dalam format berikut:
* Nama indeks vektor: "MOLSEARCH_TEST".
* Kunci: ID unik struktur molekul, seperti "168000001".
* Informasi fitur: informasi vektor berdimensi 512.
* "smiles": Formula kimia, seperti "CCC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1ccc(OC)cc1OC".
"""
num = 0
lines = []
with open(file_path, 'r') as f:
for line in f:
if line.find("smiles") >= 0:
continue
lines.append(line)
if len(lines) >= 10:
parallel_submit_lines(lines)
num += len(lines)
lines.clear()
if num % 10000 == 0:
print("load num", num)
if len(lines) > 0:
parallel_submit_lines(lines)
print("load done")
def parallel_submit_lines(lines):
"""
Metode penjadwalan untuk penulisan konkuren.
"""
with ThreadPoolExecutor(len(lines)) as t:
for line in lines:
t.submit(handle_line, line=line)
def handle_line(line):
"""
Menangani penulisan satu struktur molekul.
"""
if line.find("smiles") >= 0:
return
parts = line.strip().split(',')
try:
ids = parts[1]
smiles = parts[0]
vec = smiles_to_vector(smiles)
insert_data(ids, smiles, vec)
except Exception as result:
print(result)
def smiles_to_vector(smiles):
"""
Mengekstrak fitur vektor struktur molekul dan mengonversinya dari format SMILES ke vektor.
"""
mols = Chem.MolFromSmiles(smiles)
fp = AllChem.GetMorganFingerprintAsBitVect(mols, 2, 512 * 8)
hex_fp = DataStructs.BitVectToFPSText(fp)
vec = list(bytearray.fromhex(hex_fp))
return vec
def insert_data(id, smiles, vector):
"""
Menulis vektor struktur molekul ke Tair Vector.
"""
attr = {'smiles': smiles}
tair.tvs_hset(INDEX_NAME, id, vector, **attr)
def do_search(search_smiles,k):
"""
Masukkan struktur molekul yang akan dikueri. Metode ini mengkueri indeks tertentu di Tair dan mengembalikan k struktur molekul paling mirip.
Pertama, ekstrak fitur vektor struktur kueri. Kemudian, gunakan perintah TVS.KNNSEARCH untuk menemukan ID unik k struktur molekul terdekat (k=10 dalam contoh ini). Terakhir, gunakan perintah TVS.HMGET untuk mengambil formula kimia yang sesuai.
"""
vector = smiles_to_vector(search_smiles)
result = tair.tvs_knnsearch(INDEX_NAME, k, vector)
print("10 struktur molekul paling mirip dengan target kueri adalah sebagai berikut:")
for key, value in result:
similar_smiles = tair.tvs_hmget(INDEX_NAME, key, "smiles")
print(key, value, similar_smiles)
if __name__ == "__main__":
# Hubungkan ke database Tair dan buat indeks vektor untuk struktur molekul bernama "MOLSEARCH_TEST".
tair = get_tair()
INDEX_NAME = "MOLSEARCH_TEST"
create_index()
# Tulis data sampel.
do_load("D:\Test\Compound_168000001_168500000.smi")
# Dalam indeks MOLSEARCH_TEST, kueri 10 struktur molekul paling mirip dengan "CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1".
do_search("CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1",10)Output contoh berikut menunjukkan bahwa kode berhasil dieksekusi:
create index done
load num 10000
load done
10 struktur molekul paling mirip dengan target kueri adalah sebagai berikut:
b'168000009' 0.0 ['CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1']
b'168003114' 29534.0 ['Cc1cc(C)cc(N2CCN(CC(=O)NC3CCCC3)C(=O)C2=O)c1']
b'168000210' 60222.0 ['COc1ccc(N2CCN(CC(=O)Nc3cc(C)cc(C)c3)C(=O)C2=O)cc1OC']
b'168001000' 61123.0 ['COc1ccc(N2CCN(CC(=O)Nc3ccc(C)cc3)C(=O)C2=O)cc1OC']
b'168003038' 64524.0 ['CCN1CCN(c2cc(C)cc(C)c2)C(=O)C1=O']
b'168003095' 67591.0 ['O=C(CN1CCN(c2cccc(Cl)c2)C(=O)C1=O)NC1CCCC1']
b'168000396' 70376.0 ['COc1ccc(N2CCN(Cc3ccc(C)cc3)C(=O)C2=O)cc1OC']
b'168002227' 71121.0 ['CCOC(=O)CN1CCN(C2CC2)C(=O)C1=O']
b'168000441' 73197.0 ['Cc1cc(C)cc(NC(=O)CN2CCN(c3ccc(F)c(F)c3)C(=O)C2=O)c1']
b'168000561' 73269.0 ['Cc1cc(C)cc(N2CCN(CC(=O)Nc3ccc(C)cc3C)C(=O)C2=O)c1']Hasil
Anda juga dapat memplot struktur molekul yang mirip sebagai citra. Gambar berikut menunjukkan contohnya.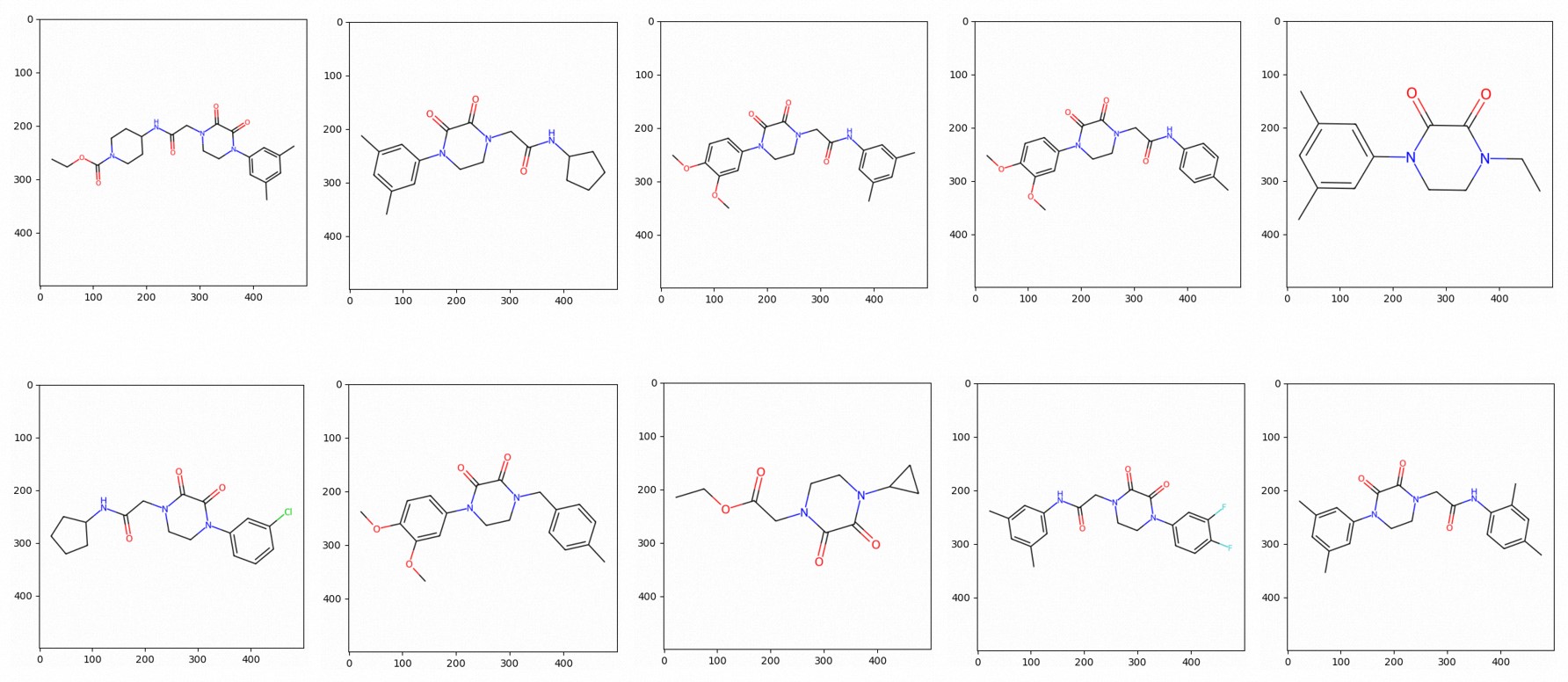
Ringkasan
Menggunakan TairVector untuk mencari struktur molekul memungkinkan Anda mengambil daftar struktur paling mirip dalam hitungan milidetik. Seiring semakin banyak set data struktur molekul disimpan di database Tair, kueri berikutnya menjadi lebih akurat dan tepat waktu. Solusi ini mengurangi waktu pengembangan dan meningkatkan efisiensi keseluruhan di bidang penemuan obat.